VMDでRMSDを計算する (GUI, CUI)
概要
vmdでのRMSDの計算する方法をまとめる。今回は水素以外のタンパク質でアラインし、タンパク質の主鎖のRMSDを計算する。0フレーム目を基準としてそれぞれ計算する。
環境
macOS Mojave 10.14.5
vmd 1.9.3
macOS Catalinaではアプリが64bitにしか対応していないためVMDが利用できない
GUIでの方法
まずは構造ファイルとトラジェクトリ ファイルを読み込む。読み込み方はこちら → VMDの使い方の基本 (Mac)
タンパク質でアライン
VMD MainのExtensions=>Analysis=>RMSD Trajectory Tools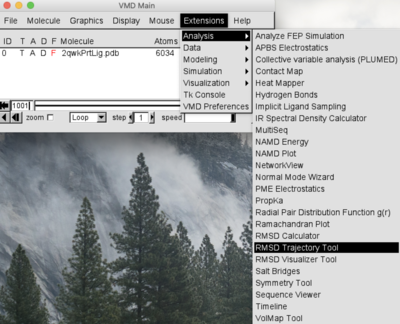
RMSD Trajectory Toolメニューの空白部分でアラインする原子を選択する。図のようにproteinとしSelection Modifiersのnohにチェックをつけるか、protein and nohとする。その後、ALIGNをクリックする。
RMSDの計算
RMSEの計算をするために、タンパク質の主鎖を指定する。図のように
proteinとしSelection ModifiersのBackboneにチェックを入れるか、protein and name CA C Nとする。さらにTrajectoryのPlotにチェックを入れRMSDをクリックする。
プロットが現れる。
FileからExport to PostScriptを選択すると画像を保存できる。また、Export to ASCII matrix...を選択すると計算結果データを保存できる。
CUIでの方法
vmdのスクリプト作成が初めてという方はこちら→VMDのスクリプトの書き方。
今回はtest.pdbとtest.dcdを読み込んでアライン、RMSDを計算する。その後、RMSDの計算結果をtest.datに保存するスクリプトを示す。
mol load pdb test.pdb xtc test.xtc set out_file [open test.dat w] set num_frame [molinfo top get numframes] set ref_fit [atomselect top "protein and noh" frame 0] set ref_rmsd [atomselect top "protein and name CA C N" frame 0] puts $out_file "frame\trmsd" for {set i 0} {$i < $num_frame} {incr i} { set all [atomselect top all frame $i] set prt_fit [atomselect top "protein and noh" frame $i] $all move [measure fit $prt_fit $ref_fit] set prt_rmsd [atomselect top "protein and name CA C N" frame $i] set rmsd [measure rmsd $prt_rmsd $ref_rmsd] puts $out_file "$i\t$rmsd" } exit
スクリプトの説明
- 構造、トラジェクトリファイルを読み込む
- アウトプットファイルを定義する
- トラジェクトリファイルのフレーム数を取り出す
- 一番最初のフレームのタンパク質情報を取り出す(このタンパク質に合わせて各フレームをアラインする)
- 一番最初のフレームのタンパク質情報を取り出す(このタンパク質に合わせて各フレームのRMSDを計算する)
- 各フレームの全原子をタンパク質でアラインする
- 各フレームのタンパク質のRMSDを計算する