VMDの使い方の基本 (Mac)
概要
VMD (Visual Molecular Dynamics) とは分子構造、シミュレーションの可視化ソフト。無料で手に入る。無料の可視化ソフトは他にもPyMolがあるので使い分けられるとよい。個人的にはPyMolの方がGUI機能が豊富だが、シミュレーション等のトラジェクトリを見る時はVMDの方が軽い印象。
環境
macOS Mojave 10.14.5
vmd 1.9.3
macOS Catalinaではアプリが64bitにしか対応していないためVMDが利用できない
インストール
公式からダウンロードする。ダウンロードする際には登録が必要となる。ダウンロードしたらそのままApplicationフォルダにつっこむと使えるようになる。
起動の仕方
起動の仕方はアプリのアイコンをクリックする方法 (GUI) とターミナルから起動する方法 (CUI) の2つがある。linuxコマンドが不自由なく使えるのであれば後者をおすすめする。
ファイルの読み込み方
GUI
VMDアプリを起動し、
File==>New Molecule ...とクリックする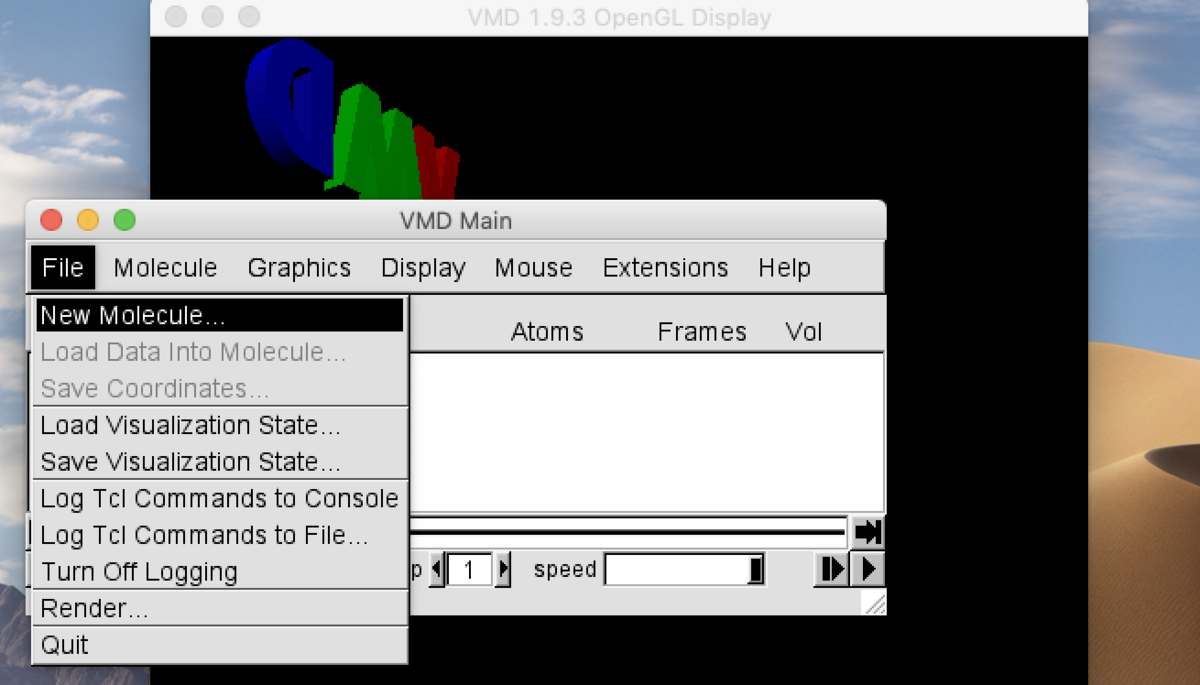
Load files forがNew Moleculeになっていることを確認する
Browse...をクリックし表示したいファイルを選ぶLoadをクリック
さらにトラジェクトリを読み込みたい時は
Load files forで先ほど読み込んだファイル名を指定し、同じようにBrowse...からのLoadをクリックする。読み込むのに少し時間がかかる。
CUI
1 . パスを通す。~/.bash_profile または~/.bashrcに以下を記入する
export PATH="/Applications/VMD 1.9.3.app/Contents/vmd/:$PATH" alias vmd='vmd_MACOSXX86'
2 . シェルを再起動する。
$ exec "$SHELL" -l
3 . 表示したいファイルのあるディレクトリへ移動
4 . vmdを起動しつつfile.pdbを読み込む
$ vmd file.pdb
5 . 同時にトラジェクトリであるfile.dcdを読み込む
$ vmd file.pdb file.dcd
グラフィック操作
VMD MainディスプレイのGraphics ==> Representations...

そうすると以下のようなディスプレイが表示される。

このディスプレイをメインに使っていく。今はStyleがLines、ColorがName、Selectionがallになっている。それぞれ以下の項目を変更することで変更できる。
- Style :
Drawing Method - Color :
Coloring Method - Selection :
Selected Atoms
Styleの変更
Drawing Methodを変更する。おすすめはタンパク質であればNew Carton。

リガンドはBonds。

Colorの変更
Coloring Methodを変更する。こちらはデフォルトでも十分見やすい。自分の指定した色にする場合はColorIDに設定する。
Selectionの変更
ここが一番大事。Selected Atoms欄にコマンドを入力してEnterを押すことで表示が切り替わる。ここは習うより慣れろということで具体例を示していく。
- タンパク質の水素以外 :
protein and noh - 水以外 :
not water - 残基名がアルギニン :
resname ARG - 残基番号が5-8 :
resid 1 to 5 - 原子名が
NまたはCA:name N CA - 残基名が
G36から5Å以内にあるアミノ酸残基 :amino within 5 of resname G39
Repositoryの追加
レポジトリを増やすと違う表示方法を同時に見比べることができる。例えば、タンパク質の残基番号が1-10番の原子だけを赤色にしたい場合、デフォルトのレポジトリと残基番号が1-10番で赤色のレポジトリを新たに作成すればよい。具体的には以下のようにする。
Create Repをクリック。これでレポジトリが2つになる。ColorをColorIDの1に設定する。Selected Atomsでresid 1 to 10とコマンドを打ってEnter

